Um genoma é um projeto genético que determina as características de um organismo. O ácido desoxirribonucléico (DNA) e, geralmente, no caso dos vírus, o ácido ribonucléico (RNA) são os blocos de construção das sequências genômicas. E a manipulação direta desses ácidos nucléicos pode levar a mudanças tangíveis no organismo.
Como tal, o desenvolvimento da engenharia genética concentra-se em nossa capacidade de manipular sequências genômicas. Mas esta é uma tarefa difícil. Por exemplo, o controle preciso de uma classe específica de moléculas de RNA projetadas, chamadas de “chaves de apoio”, pode fornecer uma visão vital dos ambientes celulares e de doenças potenciais. No entanto, experimentos anteriores mostraram que os toehold switches não são tratáveis, muitos não respondem às modificações, embora tenham sido projetados para produzir a saída desejada em resposta a uma determinada entrada com base em regras de dobramento de RNA conhecidas.
Diante disso, duas equipes de pesquisadores do Wyss Institute na Harvard University e no MIT desenvolveram um conjunto de algoritmos de aprendizado de máquina que podem melhorar esse processo. Especificamente, eles usaram o aprendizado profundo para analisar um grande volume de sequências de chaves de toehold para prever com precisão quais toeholds executam suas tarefas de forma confiável, permitindo que os pesquisadores identifiquem toeholds de alta qualidade para seus experimentos. Suas descobertas foram publicadas na Nature em dois artigos separados hoje.
Com qualquer problema de aprendizado de máquina, a primeira etapa é coletar dados específicos do domínio para treinar o modelo. Os pesquisadores coletaram um grande conjunto de dados composto de sequências de interruptores. Alex Garruss, co-primeiro autor e aluno de pós-graduação trabalhando na Wyss declarou:
Leia também:
“Projetamos e sintetizamos uma enorme biblioteca de toehold switches, quase 100.000 no total, por amostragem sistemática de regiões de gatilho curtas ao longo de todo o genoma de 23 vírus e 906 fatores de transcrição humanos.”
Como havia duas equipes diferentes, os pesquisadores tentaram duas técnicas diferentes para abordar o problema. Os autores do primeiro artigo decidiram analisar os toehold switches não como sequências de bases, mas como imagens 2D de possibilidades de pares de bases. Esta abordagem, chamada de Visualização de Mapas de Saliência de Estrutura Secundária, ou VIS4Map, identificou com sucesso os elementos físicos das chaves de apoio que influenciavam seu desempenho, fornecendo uma visão sobre os mecanismos de dobramento de RNA que não foram descobertos usando técnicas de análise tradicionais.
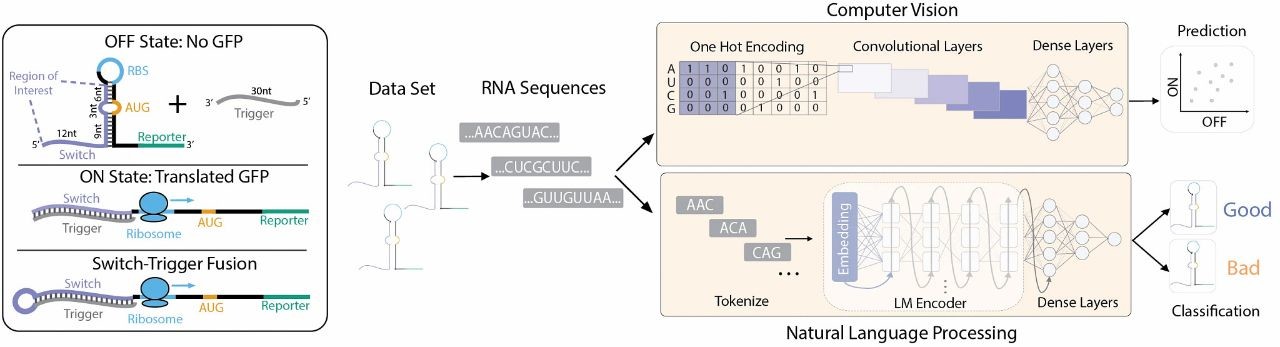
Os autores do segundo artigo criaram duas arquiteturas de aprendizado profundo diferentes que abordaram o desafio de identificar chaves de apoio de pé ‘suscetíveis’ usando técnicas ortogonais. O primeiro modelo foi baseado em rede neural convolucional (CNN) e perceptron multicamadas (MLP), que tratava as sequências de toehold como imagens 1D, ou linhas de bases de nucleotídeos. Usando uma técnica de otimização chamada Sequence-Based Toehold Optimization and Redesign Model (STORM), identificou padrões de bases e potenciais interações entre essas bases para marcar os toeholds de interesse.
A segunda arquitetura modelou o problema para o domínio do processamento de linguagem natural (PNL), tratando cada sequência de toehold como uma frase que consiste em padrões de palavras. A tarefa era treinar um modelo para combinar essas palavras, ou bases de nucleotídeos, para formar uma frase coerente. Este modelo foi integrado com o modelo baseado em CNN para criar Discurso de Ácido Nucleico (NuSpeak). Esta técnica de otimização redesenhou os últimos nove nucleotídeos de um determinado toehold switch, mantendo os 21 nucleotídeos restantes intactos. Isso permitiu a criação de suportes para pés especializados que detectam a presença de sequências de RNA patogênicas específicas e podem ser usados para desenvolver novos testes diagnósticos.
Para testar os dois modelos, os pesquisadores detectaram fragmentos do SARS-CoV-2, o genoma viral que causa o COVID-19, usando suas chaves de apoio otimizadas. NuSpeak melhorou o desempenho dos sensores em uma média de 160%. Por outro lado, o STORM criou versões melhores de quatro sensores de RNA viral SARS-CoV-2, melhorando seu desempenho em até 28 vezes. A propósito desses resultados impressionantes, a co-primeira autora do segundo artigo, Katie Collins, uma estudante do MIT no Wyss Institute, afirmou:
“Um benefício real das plataformas STORM e NuSpeak é que elas permitem que você projete e otimize rapidamente componentes de biologia sintética, como mostramos com o desenvolvimento de sensores de suporte para um diagnóstico COVID-19.”
Diogo Camacho, autor correspondente do segundo artigo e Cientista Sênior de Bioinformática e co-líder da Predictive BioAnalytics Initiative do Wyss Institute declarou:
“Talvez o aspecto mais importante das ferramentas que desenvolvemos nestes artigos é que elas são generalizáveis para outros tipos de sequências baseadas em RNA, como promotores indutíveis e riboswitches de ocorrência natural e, portanto, podem ser aplicadas a uma ampla gama de problemas e oportunidades em biotecnologia e medicina. ”
Seguindo em frente, como Camacho imaginou, as equipes estão procurando generalizar seus algoritmos para mapeá-los em outros problemas da biologia sintética para potencialmente acelerar o desenvolvimento de ferramentas de biotecnologia.